Research
Research
Carbohydrate Mass Spectrometry
● Development of new mass spectrometry method for carbohydrate structural identification
● Development of new mass spectrometer for carbohydrate structural identification
● Investigation of carbohydrate dissociation mechanism in gas phase
Nucleic acids, proteins, and carbohydrates are the three macromolecules involved in creating life. The structures of these macromolecules are complicated because of different subunits and sequence variations among these subunits. There are well-established methods for sequencing nucleobases in nucleic acids and amino acids in proteins, but the development of a robust analytical method to determine the primary structure of carbohydrates remains a challenge. The main difficulty in the primary structure determination of carbohydrates is caused by the large number of isomers. For example, the number of oligosaccharide isomers such as trisaccharide and tetrasaccharide can be as large as 3240 and 139,968, respectively even when considering only the oligosaccharides generated from three common hexoses (e.g., glucose, galactose, and mannose). The number of isomers is large and the differences between structures are subtle, making differentiation between these isomers extremely difficult.
Traditional analytical methods for structural determination of oligosaccharides use chemical reactions, or nuclear magnetic resonance (NMR) spectroscopy, both methods are time-consuming and require large amount of samples; therefore, they are not suitable for low-volume samples, such as oligosaccharides extracted from cancer cells or antibodies. The sensitivity of mass spectrometry is three to four orders of magnitude higher than the traditional chemical methods and NMR spectroscopy, and mass spectrometry is thus particularly useful for low-volume samples.
In primary structural analysis of oligosaccharides by mass spectrometry, accurate mass measurement from single-stage mass spectrometry reveals only the oligosaccharide composition. Multistage tandem mass spectrometry is necessary for structural determination. However, conventional mass spectrometry methods only provide information regarding the sequence and linkage positions in oligosaccharides. Information of anomeric configurations (α-and β-anomericity) and stereoisomers (e.g., glucose, galactose, and mannose, or GlcNAc and GalNAc) in oligosaccharides is not available from these methods.
We recently developed a new mass spectrometry-based method, namely logically derived sequence (LODES) multistage tandem mass spectrometry (MSn), for the primary oligosaccharide structural determination. LODES/MSn involves sequential low-energy CID of oligosaccharides in an ion-trap mass spectrometer, wherein the sequences of CID in MSn are derived from carbohydrate dissociation mechanisms. LODES/MSn can determine linkage positions and anomeric configurations, and differentiate stereoisomers. It has high sensitivity and can be used for real-time online structural determination of small oligosaccharides eluted from high-performance liquid chromatography (HPLC). Complete CID sequences for tandem mass spectrometry can be programmed into computers, making the automatic structural determination of oligosaccharides possible.
Current LODES/MSn can be applied to the oligosaccharides consisting of glucose, galactose, mannose, N-acetylglucosamine, N-acetylgalactosamine, xylose, arabinose, fucose, sialic acid, and fructose. Extension of LODES/MSn to the oligosaccharides consisting of other monosaccharides is currently under development.
l MALDI ionization mechanism
l Photodissociation of organic molecules in a molecular beam
l Energy transfer of highly vibrationally excited molecules studied by crossed molecular beams
研究興趣
1.醣質譜和發展新型質譜儀 2.基質輔助雷射脫附游離機制 3.分子束中的有機分子、小型生化分子的光化學 4.高能量分子的能量轉移及超級碰撞1. 醣質譜
我們的研究主題,利用過去幾年研究有機分子在分子束中光分解、分子束中分子間碰撞能量轉移的經驗,用來研究醣分子在質譜儀中的分解機制,並進而提出一個利用質譜儀來鑑定醣分子結構的新方法。
醣、蛋白質、核酸、脂質是構成生命的四大類分子。除了脂質分子因為比較小,容易鑑定其結構之外,蛋白質、核酸、和醣都是巨分子,分子結構的鑑定都非常不容易。早年英國科學家Frederick Sanger發明決定蛋白質和核酸序列的方法,也因此分別在1958年1980年獲得諾貝爾化學獎(他是目前為止,唯一獲得兩次諾貝爾化學獎的得獎者)。現在只剩下醣分子,還沒有一個簡單、可靠的辦法,可以其分子結構。
醣分子的結構非常複雜、樣品合成困難、從生物體中不易大量取得、混合物分離困難,都是造成醣分子結構非常難判斷的主要原因。目前分析醣分子的結構,主要是用核磁共振儀和質譜儀來決定。對於稍微大一點的醣分子,其結構鑑定還是有很多困難。核磁共振儀所需要的樣品量比質譜儀多非常多,因此不適合用來研究從生物體系萃取出來的少量醣分子,因此質譜儀變成研究醣結構最重要的工具。
目前用質譜儀研究醣的結構,還是遇到非常多的困難。例如靈敏度還是不足、比較複雜的醣分子就無法判斷其完整的結構等。我們利用物理化學的方法,包括高準確度的量子化學計算以及實驗,了解醣分子在質譜儀中的分解機制。藉由了解其分解機制,我們可以預測醣分子的分解途徑,進而決定醣分子的完整結構。目前我們已經發展了一個新方法,用質譜儀來決定醣分子的結構。這個方法比過去文獻中所有的方法,靈敏度高很多,速度也快非常多。我們將在這個研究方向上繼續探討,並將這個方法應用從生物體系萃取出來的少量醣分子。
2.基質輔助雷射脫附游離機制
基質輔助雷射脫附游離是一種近年來被廣泛運用於生物分子質譜上的軟游離,其全名為Matrix Assisted Laser Desorption Ionization,簡稱MALDI。它是在1980年末期,由Tanaka, Karas和Hillenkamp 共同發明,其中Tanaka也因此得到了2002年的諾貝爾化學獎。簡單來說,MALDI是將基質和分析物以一定的比例混合成固體混合物之後,將樣品放在質譜儀中,並以紫外光脈衝雷射 (通常是337nm 或 355nm)照射。由於基質分子對雷射光波長有很大的吸收係數,樣品照射了雷射光之後,吸收了雷射光,將光的能量轉換成熱,樣品被雷射光照到的部份溫度會急劇上昇。由於溫度的上升,分子就相當容易由固體轉換成氣態,形成大量的氣體,並脫離樣品表面。在這個氣體裡面除了大部分的中性分子之外,還有正離子、負離子,甚至於也有電子。將這些離子送進質譜儀,便可偵測出離子的質荷比,而得到分析物的質譜。與傳統的方法比較,因為MALDI是一種軟游離,不需要事先將分析物氣化,產生的碎片較少,可以得到分析物的原始質量,因此近年來科學家大量應用在無法氣化又易碎的生物分子上。
雖然MALDI的發明至今已超過20年,但其游離機制仍然不清楚。另外,MALDI的缺點還包括:離子訊號的忽大忽小、對於醣類分靈敏度低、訊號的再現性不佳等,這些缺點對於MALDI的應用有著極大的影響。例如:難以用MALDI做定量分析、MALDI在醣類分子的應用有限等。我們認為如果能充分瞭解MALDI的游離機制,就有機會依照游離機制的原理,提出一個改善MALDI缺點的辦法,使MALDI在應用上可以更為廣泛。
在之前的研究中,有很多種MALDI游離機制模型被提出來。但在這些模型中,大部分都因為缺乏實驗數據支持,而無法確認其是否為MALDI的游離機制。其中只有三個模型提出可供量化的數據以便和實驗比較。這三個模型包括thermal ionization of photoexcited matrix model、exciton pooling model、solid state thermal model,其中thermal ionization of photoexcited matrix model和exciton pooling model都號稱模型預測的數據和實驗數據非常接近。尤其是exciton pooling model,經由作者的大力鼓吹,導致近年來有人覺得exciton pooling model可能是對的。
最近我們改裝兩台不同性能的交叉分子束儀器,詳細測量了脫附分子中的離子-中性分子比(ion-to-neutral ratio)。我們發表文章,指出過去20年來所有理論的模型建造者,都錯把『馮京』當『馬涼』,完全誤會了過去的實驗數據。他們都錯把『ion-to-neutral ratio of analyte』當成『ion-to-neutral ratio of matrix』了。他們號稱模型預測的數據和實驗數據非常接近,實際上是和實驗數據差了10000倍,因此有必要重新檢視所有的模型。 我們利用超快瑩光光譜,證明大部分的基質分子是完全不進行exciton pooling,因此exciton pooling至少不是MALDI普遍的游離機制。另外,我們利用無溶劑法配制MALDI樣品,並和傳統溶劑法配制的MALDI樣品比較,發現離子產生的量是一樣的,這證明需要經由溶劑產生的preformed ions是不重要的。我們也測量不同雷射脈衝長度的脫附分子中的離子-中性分子比,發現它不隨不同雷射脈衝長度而改變,證明多光子游離也不是重要游離機制。
最近我們提出一個新的模型,即熱致質子轉移模型(thermal proton transfer model) ,說明熱致質子轉移反應(thermally induced proton transfer reaction) 是普遍存在MALDI游離機制中,它是非常重要的MALDI游離機制。本模型可以準確的預測脫附分子中的離子-中性分子比、可以說明不同分析物所產稱的離子量隨分析物的質子親和力而改變、也可以解釋附分子中的離子-中性分子比不隨雷射脈衝長度而改變,證明熱致質子轉移反應是最重要的游離機制。
有關利用多質量離子影像儀研究光分解的入門資料,請參考我們發表在Ann. Rev. Anal. Chem. 8, 21-39 (2015)的回顧性論文。我們最新的相關論文則有J. Am. Soc. Mass Spectrom. 27, 1491-1498 (2016)和 J. Am. Soc. Mass. Spectrom. 28, 2235-2245 (2017)。
3.分子束中有機分子、小型生化分子(胺基酸、鹼基等)的光化學
主要的研究,是利用最先端的物理化學實驗方法,包括高真空、分子束、雷射、非線性光學、離子影像技術等,來研究有機分子、小型生化分子的光化學、光物理行為。目前正在研究的,包括在分子束中,苯環衍生物、胺基酸和鹼基衍生物的光分解,光同份異構化轉換等。
我們利用本實驗室自行研發(也是全世界目前唯一)的多質量離子影像儀,如下圖,來測量分子受光後的各種行為。這個儀器可以同時測量好幾個不同碎片的質量及速度分布,效率比一般的質譜儀好很多倍,因此可以進行一些其他實驗室不易進行的實驗。這些測量,配合量子力學的計算,可以讓我們在沒有溶劑分子的干擾下,詳細研究分子吸收光子之後的反應機構。

下圖是光分解甲苯(C6H5CD3)所得的碎片離子影像。影像顯示我們可以同時測量到碎片CH3、CH2D、CHD2、CD3及C6H5、C6H4D、C6H3D2、C6H2D3等碎及其移動動能的分布。這個研究結果告訴我們:甲苯吸收光之後,甲基上的D原子會和苯環上的H原子交換,然後才進行分解。這是一個以前未曾知道的反應機構。

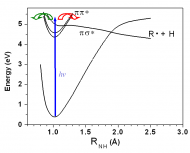
我們最近研究生化分子的光化學,也發現一些很有趣的現象。在光化學上,生化分子有一個共同的特點:即紫外光的螢光量子產率都非常低。一般認為這是和這些分子的快速內轉換(internal conversion)有關。這些分子在吸收紫外光之後,很快將電子激發態之電子能量經由內轉換轉移成電子基態之振動能,再把多餘的振動能傳遞至周圍的溶液分子,然後恢復最初的狀態。如左圖綠色箭頭所示。這也是大家認為這些生化分子具光穩定性(即不因照光而進行化學反應)的主要原因。最近德國的理論學家則經由計算提出另外一個反應機構:即這些分子在吸收紫外光之後,其中一個氫原子很快在電子激發態經由排斥性的位能曲面而脫離。如圖紅色箭頭所示。如果是這樣,那麼因氫原子脫離而產生的自由基,將有很大的機會與周圍溶液分子反應,並破壞這些生化分子的光穩定性。我們利用多質量離子影像技術來研究這些分子可能的反應機構,發現當分子比胺基酸小時,確是如理論計算的預測。但當分子比胺基酸大時,分子只進行快速的內轉換,將激發態的電子能量快速得地轉變成振動能,然後恢復最初的狀態。
有關利用多質量離子影像儀研究光分解的入門資料,請參考我們發表在International Review in Physical Chemistry, 23, 187 (2004)及J. Phys. Chem. B 111, 12631 (2007 invited feature article)的兩篇回顧性文章。有關生化分子光穩定性的文章,請參考我們發表的文章J. Chem. Phys. 126, 241104 (2007)、Phys. Chem. Chem. Phys. 12, 4989 (2010).
4.高能量分子的能量轉移及超級碰撞
能量在化學反應中佔有很重要的地位,分子一定要有足夠的能量克服化學反應中的位能障礙,才能產生化學變化。十九世紀末,二十世紀初,科學家發現很多儲存在容器中的化合物會因為化學鍵斷裂而產生分解反應。但是,困擾化學家的是:造成化學鍵斷裂所需要的能量,是從哪裡來的呢?西元一九一九年,Perrin提出一個假說:分子是吸收容器器壁發射的輻射光而得到足夠能量,之後才進行反應。但是Langmuir則指出,若要使分子得到足夠能量而進行反應,容器必須放出波長小於400nm的光讓分子吸收,然而即使容器到了700℃,根據黑體輻射的計算,器壁放出此波長的光的機率機乎等於零,所以這個吸收輻射光的假說是不成立的。西元一九二二年,Lindemann和Hinshelwood提出另一個理論:容器中的分子原本皆處於基態(能量很低),分子之間會藉由碰撞,產生能量轉移的情形。若分子經由碰撞而累積得到超過化學反應所需的能量,它就會進行反應。

雖然Lindemann 和Hinshelwood 的理論能夠說明反應速率隨壓力減少而變慢的現象,但是實際上,這個理論所得的預測值卻和實驗值相差很大。科學家發現這個理論基本上是假設分子之間的碰撞是「強烈碰撞」(strong collision),也就是分子碰撞時所轉移能量的大小,必須能夠隨時補充高能量分子因反應而減少的數目,讓分子的能量維持常態的波茲曼分佈(Boltzmann distribution)。很可惜地,分子能量並不會一直維持波滋曼分佈,因為實際上分子之間的碰撞大部分都是「微弱碰撞」(weak collision),那就是藉由碰撞所轉移的能量非常有限,無法維持波滋曼分佈。分子間碰撞能量轉移事實上是有大有小的,科學家用方程式來表達,分子由初始能量轉E’變為最後能量E的機率:P(E,E'),稱為「能量轉移機率分佈函數」。之後人們做實驗、從事研究工作,都是要找出真正的P(E,E'),以便了解能量轉移的機制。更特別的是:一九八0年代科學家提出「超級碰撞」(supercollisions)的概念:分子之間單一的碰撞有可能轉移非常大量的能量。雖然超級碰撞的機率很小,但是因為只要一次的碰撞,就可以轉移非常多的能量,它對化學反應的影響是非常大的。例如模擬計算指出: 1%的超級碰撞,可以讓整個反應的速率增加3至11倍。不過,真的有超級碰撞嗎?如果有,那麼是經由什麼樣的機制來達成一次碰撞就轉移巨大的能量呢?
幾十年來,科學家們嘗試各種方法,希望能求出能量轉移機率分佈函數P(E,E')。主要的方法是透過時間解析的紫外光吸收(UVA)、紅外光螢光(IRF) 、紅外光吸收、選擇性游離等方法,觀察高能量分子的能量隨著時間(碰撞次數)遞減的情形而得到轉移能量的平均值。不幸的是,這些實驗所得到的結果都是分子經過多次碰撞之後的平均轉移能量,而不是真正的P(E,E')。雖然從這些實驗提供的平均值,證實了先前為了修正Lindemann-Hinshelwood理論所做的假設,也就是大部分分子之間的碰撞是微弱碰撞。但是這些平均值卻卻無法告訴我們是否有超級碰撞,也無法提供足夠的訊息來瞭解分子間能量轉移的機制。
自二○○四年,我們研究團隊開始思考並設計一套儀器,目的就是可以直接用實驗的方式得到P(E,E'),並且也可以直接觀察超級碰撞的存在與否。首先,我們希望所做的實驗是單一碰撞的,而不是數次、數十次、甚至數百次碰撞之後的平均值,這樣很多微小的訊息才不會都被平均的動作所掩蓋了。最重要的是:這樣才能直接測量P(E,E')並觀察超級碰撞。我們設計並組裝交叉分子束及先進的時間切片離子影像儀來研究分子間的能量轉移。
過去也曾經有人用交叉分子束來研究分子間的能量轉移,但是一直沒有成功。重要的原因,是很難產生高純度的高能量分子束,因此實驗的背景雜訊太大而掩蓋了真正的訊號。這種實驗遇到困難的情形,就像是想要量船長的體重,用的方法卻是先秤船艦和船長的總重,再減去船艦的重量一樣。因為船艦的重量太大了,測量船艦重量所產生的誤差,就已經遠遠大於船長本身的重量,因此這個方法是無法測出船長的體重。但是如果我們能夠把船艦的重量變得很小,例如變成像一個玩具小船一樣重。那麼用同樣的方法,先秤出玩具船和船長的總重,再減去玩具船的重量,是可以得到準確的船長體重。因此我們研究的第一個重點,就是如何產生高純度的高能量分子束,讓其中不具高能量的分子濃度降到最低。我們發展了利用真空紫外光雷射來測量分子束中高能量分子濃度的方法,並且經過長期地搜尋,我們很幸運地找到可以利用薁分子來產生很高濃度的高能量分子束,並且利用它來研究高能量分子和鈍氣原子之間的能量轉移。
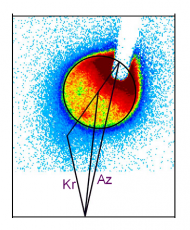
上圖為高能量薁與氪原子在交叉分子束中碰撞的散射影像結果,從這影像中,我們不但可以直接得到P(E,E'),也可以發現一些有趣的現象。例如我們發現的確有類似超級碰撞的現象,也就是一次碰撞轉移很多的能量,但是這種現象,大部分只發生在“近距離”的碰撞。另外,少部分高能量薁分子和氪原子在碰撞過程中,形成短暫的凡得瓦分子,雖然在形成凡得瓦分子的過程中,高能量薁分子和氪原子相處在一起的時間很長,可是能量卻不會從能量多的薁分子流向氪原子。相反地,是將氪原子的動能流向薁分子的轉動及振動內能。這些有趣的現象,都是當初始料未及。

